- 首頁
- 資訊
JACS:DNA折紙助力RBD納米排布對新冠感染和免疫激活影響的研究
來源:廈門大學化學化工學院 2022-11-16
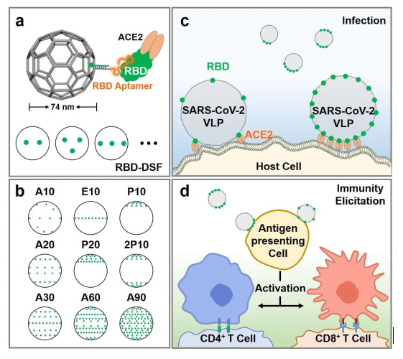
導讀:近日,廈門大學化學化工學院宋彥齡教授和上海交通大學楊洋研究員合作,在新冠病毒感染機制研究領域取得新進展,相關成果以"Elucidating the effect of nanoscale receptor-binding domain organization on SARS-CoV-2 infection and immunity activation with DNA origami"為題,發表于《美國化學會志》(J. Am. Chem .Soc. DOI:10.1021/jacs.2c09229)。
自新冠肺炎疫情暴發以來,宋彥齡研究組致力于研發中和適體以阻斷新冠病毒侵染,并取得了系列研究進展(Anal. Chem. 2020, 92 (14), 9895-9900;Angew. Chem., Int. Ed. 2021, 60 (18), 10266-10272;J. Am. Chem. Soc. 2021, 143 (51), 21541–21548;2022, 144 (29), 13146-13153; ACS Nano 2022, 16 (9), 15310?15317)。自然界存在豐富的多價受體-配體相互作用,不同病毒在配體數量和拓撲分布展現出不同特征,以平衡侵染效率和能量消耗。以包膜病毒為例,其與宿主細胞通過多價作用實現病毒侵染。然而,不同包膜病毒在刺突蛋白(Spike)分布(數量、間距、幾何圖案)上呈現顯著差異,從而導致不同的感染特性。例如,SARS-CoV-2上的Spike蛋白/受體結合域(RBD)分布遠比流感病毒的稀疏,卻能引起更為嚴重且持續的全球疫情大流行。盡管結構生物學家已經獲得了單個Spike/RBD-ACE2的晶體結構,然而由RBD的數量和分布模式引起的病毒侵染和免疫刺激反應的差異尚不清楚。因此,亟需獲得一個具有空間精確抗原排布的類病毒顆粒,以探究具有不同抗原個數和分布模式的病毒是如何實現高效感染和免疫激活。為此,該工作通過DNA折紙技術構建了具有空間精確RBD排布的SARS-CoV-2病毒樣顆粒(SARS-CoV-2 VLP)以模擬天然病毒。為了保留更多的RBD蛋白上的活性位點,采取團隊前期發展的靶向RBD非ACE2結合區域的CoV2-4C核酸適體,從而更準確地模擬病毒-宿主細胞的相互作用和免疫反應。首先,通過CoV2-4C核酸適體引導,將RBD排布在直徑約74 nm的DNA足球框架上,進行SARS-CoV-2 VLP和受體細胞的結合動力學、熱力學性質的系統分析。研究發現,SARS-CoV-2 VLP和宿主細胞結合的親和力、結合速率隨著RBD數目的增加而增加;RBD集中分布比均勻分布具有與宿主細胞更強、更快的結合熱力學和動力學特征。此外,免疫激活程度并不隨著RBD個數增加而線性增加。每個SARS-CoV-2 VLP上僅需20個均勻分布的RBD就能實現巨噬細胞高達86%的免疫激活。總之,這項工作為研究SARS-CoV-2感染機制和類病毒顆粒疫苗的免疫激活提供了一個新工具。該工作在廈門大學化學化工學院宋彥齡教授和上海交通大學楊洋研究員的共同指導下完成。我院2021級博士生張佳露和上海交通大學醫學院附屬仁濟醫院檢驗科博士后徐蕓蕓為論文的共同第一作者。我院楊朝勇教授為該研究提供了寶貴的意見。研究工作得到國家自然科學基金項目(22022409、21735004、21874089、21977069)等資助。參考資料:https://chem.xmu.edu.cn/info/1274/16854.htm
聲明:化學加刊發或者轉載此文只是出于傳遞、分享更多信息之目的,并不意味認同其觀點或證實其描述。若有來源標注錯誤或侵犯了您的合法權益,請作者持權屬證明與本網聯系,我們將及時更正、刪除,謝謝。 電話:18676881059,郵箱:gongjian@huaxuejia.cn