


2021年7月16日,清華大學藥學院胡澤平課題組在《分析化學》(Analytical Chemistry)期刊在線發表題為《基于3-硝基苯肼同步衍生化羰基、羧基和磷酸基亞代謝組策略的超靈敏、寬覆蓋LC-MS/MS靶向代謝組學方法》(Simultaneous 3-Nitrophenylhydrazine Derivatization Strategy of Carbonyl, Carboxyl and Phosphoryl Submetabolome for LC-MS/MS Based Targeted Metabolomics with Improved Sensitivity and Coverage)的文章,通過采用 3-NPH衍生化策略,建立了基于 LC-MS/MS 的代謝組學分析方法,該方法同時靶向含羰基、羧基和磷酸基的代謝物,具有超高的檢測靈敏度和廣泛的代謝物覆蓋范圍。此外,該方法還可以簡化細胞中糖酵解、三羧酸循環和戊糖磷酸途徑的代謝流分析。代謝是生命活動的基礎,代謝異常與許多疾病的發生發展有著密切的關系。代謝組學是闡明代謝調控和揭示疾病機制的有力工具,在理解疾病機制、發現治療靶標及精準醫療中發揮了重要作用。
盡管近年來代謝組學技術已經取得了顯著進步,但仍然存在許多技術挑戰需要解決【1-3】。當前的代謝組學方法仍不夠靈敏,對代謝組的覆蓋度也非常有限,尤其是在分析微量生物樣本時,例如干細胞、胚胎發育樣本等。因此,仍需要進一步提高代謝組學分析方法的靈敏度,以在更少量的生物樣本中檢測到更多的代謝物。但是,代謝物理化性質的多樣性使得開發全面覆蓋整個代謝組的方法變得非常困難【4,5】。
化學衍生化是提高代謝物檢測靈敏度的有效手段【6-8】。然而,一種衍生化試劑通常只能與一種特定的化學基團反應【9,10】,導致目前基于衍生化的代謝組學分析技術對代謝組的覆蓋范圍非常有限。因此,開發一種可以同時覆蓋多種功能基團的衍生化策略將對代謝組學分析大有裨益。
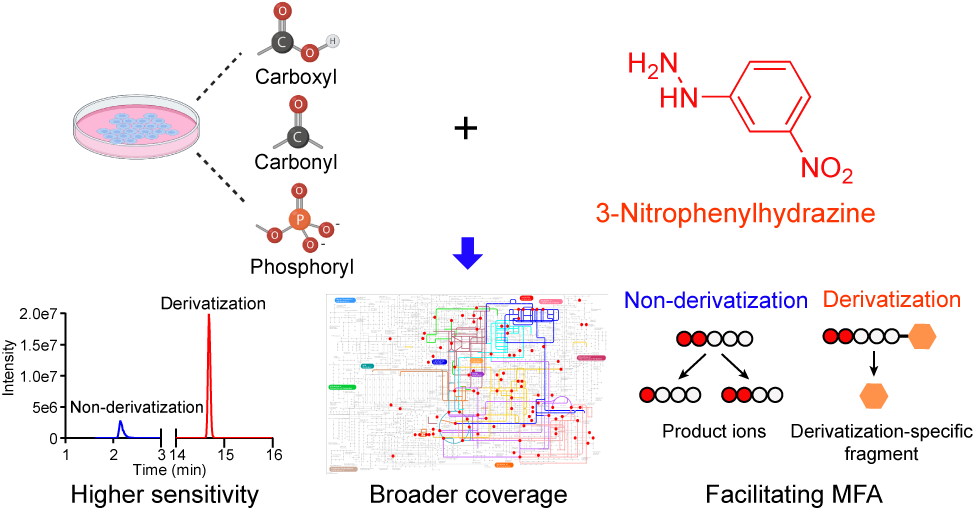
圖1. 3-硝基苯肼衍生化策略提高代謝組檢測靈敏度、拓寬覆蓋度、簡化代謝流分析
在本研究中,作者采用3-硝基苯肼(3-nitrophenylhydrazine, 3-NPH)作為衍生化試劑,對羰基代謝物、羧基代謝物和磷酸基代謝物進行同步衍生化,從而使用一種衍生試劑同時衍生化多種類代謝物,極大地提高了單次分析中代謝物的覆蓋度。
該方法共涵蓋了130種代謝物,包括了有機酸、氨基酸、糖類、核苷酸、肉堿和維生素等。通過與非衍生化方法的靈敏度進行比較,采用該衍生化方法,近 2/3 的代謝物靈敏度達到了2-300倍的提高。
同時,衍生化可以將極性代謝物轉化為其疏水性衍生物,從而增強代謝物在反相色譜柱上的色譜保留行為,提高了該方法的色譜分離能力,使得多種難以區分的同分異構體通過該衍生化方法實現了良好的分離。值得一提的是,本研究首次發現3-NPH可作為磷酸基代謝物的衍生化試劑,這為該類代謝物的全面分析和拓寬代謝組學覆蓋度提供了重要工具。
為了考察該方法在少量生物樣品中的適用性,作者將該方法應用于 60 個小鼠卵母細胞和 5000 個小鼠造血干細胞(hematopoietic stem cell, HSC)的代謝組學分析。
在60個卵母細胞中,該方法檢測到了72種代謝物,并利用該方法獲得的代謝組學數據很好地區分了卵母細胞兩個發育階段(GV期和MII期)之間代謝差異。在5000個小鼠HSC中,該衍生化方法檢測到了62種代謝物,比采用非衍生化方法多檢測到了17種代謝物,這其中包含了葡萄糖-6-磷酸、甘油醛-3-磷酸、異檸檬酸、α-酮戊二酸等多種具有重要生物學功能的代謝物。
此外,在該研究中,作者發現糖酵解、三羧酸循環和戊糖磷酸途徑中的許多代謝物在衍生化后可產生高強度的來源于衍生化試劑的質譜裂解碎片,采用該裂解碎片進行代謝流分析,大大減少了代謝流分析中需要監測的代謝物離子對的數量,并且避免了推斷代謝物質譜碎裂行為的繁瑣工作,從而簡化了代謝流分析流程。
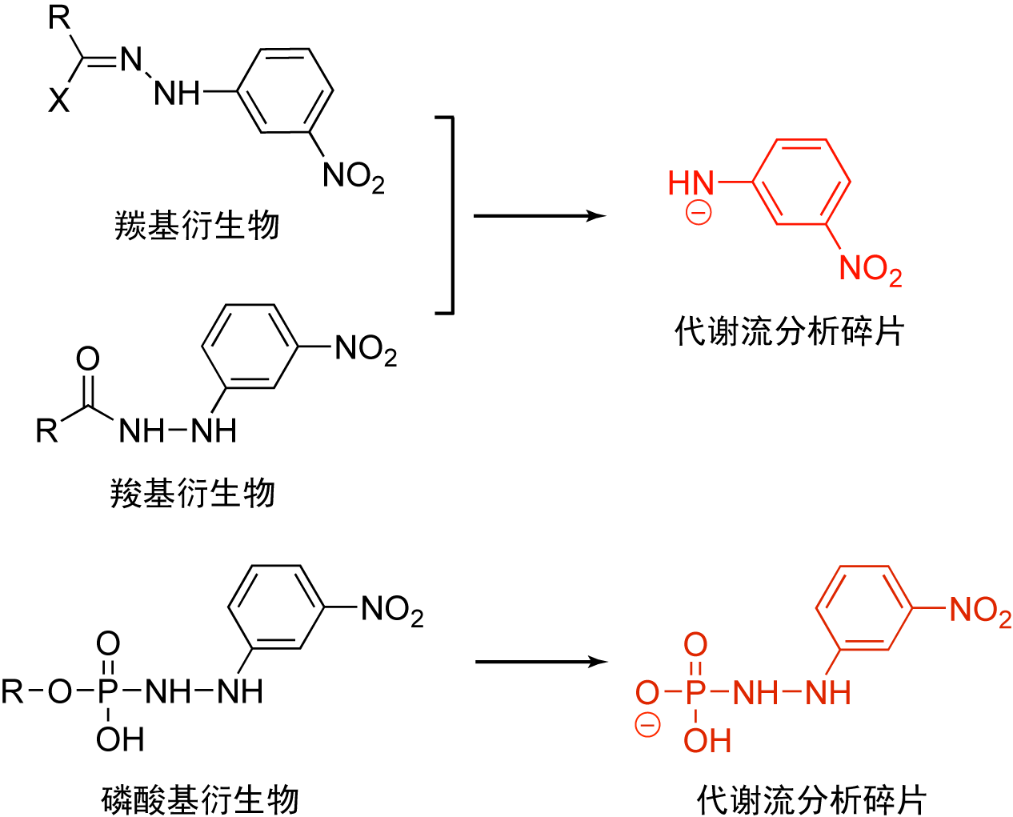
圖2. 代謝流分析監測的衍生物碎片離子結構圖
綜上,這種基于 3-NPH 衍生化的新方法具有極高的檢測靈敏度、代謝物覆蓋度和良好的色譜分離度,并且能夠簡化代謝流分析。該方法有望廣泛應用于多種代謝研究,尤其是針對微量生物樣本的代謝研究。
清華大學藥學院博士博士后孟祥駿、龐歡歡為該論文的共同第一作者。清華大學藥學院胡澤平研究員為通訊作者。首都醫科大學李兵輝教授、清華大學頡偉、王建偉等教授對該研究提供了大力支持和幫助。該研究工作得到了國家自然科學基金委“糖脂代謝的時空網絡調控”重大計劃的重點項目、科技部“發育編程及其代謝調節”重點專項、清華-北大聯合生命中心等項目支持。
原文鏈接:https://pubs.acs.org/doi/10.1021/acs.analchem.1c00767
參考文獻:
(1) Li, Y.; Bouza, M.; Wu, C.; Guo, H.; Huang, D.; Doron, G.; Temenoff, J. S.; Stecenko, A. A.; Wang, Z. L.; Fernández, F. M. Sub-nanoliter metabolomics via mass spectrometry to characterize volume-limited samples. Nat. Commun. 2020, 11, 5625.
(2) Newgard, C. B. Metabolomics and Metabolic Diseases: Where Do We Stand? Cell Metab. 2017, 25, 43-56.
(3) Agathocleous, M.; Meacham, C. E.; Burgess, R. J.; Piskounova, E.; Zhao, Z.; Crane, G. M.; Cowin, B. L.; Bruner, E.; Murphy, M. M.; Chen, W.; Spangrude, G. J.; Hu, Z.; DeBerardinis, R. J.; Morrison, S. J. Ascorbate regulates haematopoietic stem cell function and leukaemogenesis. Nature 2017, 549, 476-481.
(4) Chen, Y.; Zhou, Z.; Yang, W.; Bi, N.; Xu, J.; He, J.; Zhang, R.; Wang, L.; Abliz, Z. Development of a Data-Independent Targeted Metabolomics Method for Relative Quantification Using Liquid Chromatography Coupled with Tandem Mass Spectrometry. Anal. Chem. 2017, 89, 6954-6962.
(5) Zhao, S.; Li, H.; Han, W.; Chan, W.; Li, L. Metabolomic Coverage of Chemical-Group-Submetabolome Analysis: Group Classification and Four-Channel Chemical Isotope Labeling LC-MS. Anal. Chem. 2019, 91, 12108-12115.
(6) Vuckovic, D. Improving metabolome coverage and data quality: advancing metabolomics and lipidomics for biomarker discovery. Chem. Commun. (Camb.) 2018, 54, 6728-6749.
(7) Higashi, T.; Ogawa, S. Derivatization-based sample-multiplexing for enhancing throughput in liquid chromatography/tandem mass spectrometry quantification of metabolites: an overview. J. Chromatogr. A 2020, 1634, 461679.
(8) Zhang, H.; Shi, X.; Vu, N. Q.; Li, G.; Li, Z.; Shi, Y.; Li, M.; Wang, B.; Welham, N. V.; Patankar, M. S.; Weisman, P.; Li, L. On-Tissue Derivatization with Girard's Reagent P Enhances N-Glycan Signals for Formalin-Fixed Paraffin-Embedded Tissue Sections in MALDI Mass Spectrometry Imaging. Anal. Chem. 2020, 92, 13361-13368.
(9) Zhao, S.; Li, L. Dansylhydrazine Isotope Labeling LC-MS for Comprehensive Carboxylic Acid Submetabolome Profiling. Anal. Chem. 2018, 90, 13514-13522.
(10) Zhao, S.; Dawe, M.; Guo, K.; Li, L. Development of High-Performance Chemical Isotope Labeling LC-MS for Profiling the Carbonyl Submetabolome. Anal. Chem. 2017, 89, 6758-6765.
聲明:化學加刊發或者轉載此文只是出于傳遞、分享更多信息之目的,并不意味認同其觀點或證實其描述。若有來源標注錯誤或侵犯了您的合法權益,請作者持權屬證明與本網聯系,我們將及時更正、刪除,謝謝。 電話:18676881059,郵箱:gongjian@huaxuejia.cn